傻瓜系列教程断断续续出了5篇了,依次为:
- 生信宝典之傻瓜式 (一) 如何提取指定位置的基因组序列
- 生信宝典之傻瓜式 (二) 如何快速查找指定基因的调控网络
- 生信宝典之傻瓜式 (三) 我的基因在哪里发光 - 如何查找基因在发表研究中的表达
- 生信宝典之傻瓜式 (四) 蛋白蛋白互作网络在线搜索
- 生信宝典之傻瓜式 (五) 文献挖掘查找指定基因调控网络
这次介绍的是如何查找某个转录因子在某个细胞系中调控哪些基因。
转录因子对基因的调控最直接的方式就是从转录因子是否在对应基因启动子区或邻近有结合,这是TF ChIP-seq所要解决的问题 (当然ATAC-seq配合motif分析也可以)。
哈佛大学刘小乐教授课题组收集并重头分析了38,667个TF、Histone、Chromatin Accessibility数据并把每个样品的质量评估、比对结果、峰图数据、结合peak和靶基因等数据共享在CISTROME平台供我们使用。
这样我们在预实验和确定研究思路时不需要把数据下载下来、分析一遍,而是直接在平台搜索、点选即可获得结果。
CISTROME访问链接是http://cistrome.org/db/ (微信中不允许外链,不想拷贝输入的可以后来回复 cistrome获取可点击的链接)。
操作很简单,官网还提供了一个9分钟视频,练练英语也不错。
搜索感兴趣的细胞系或TF、Histone
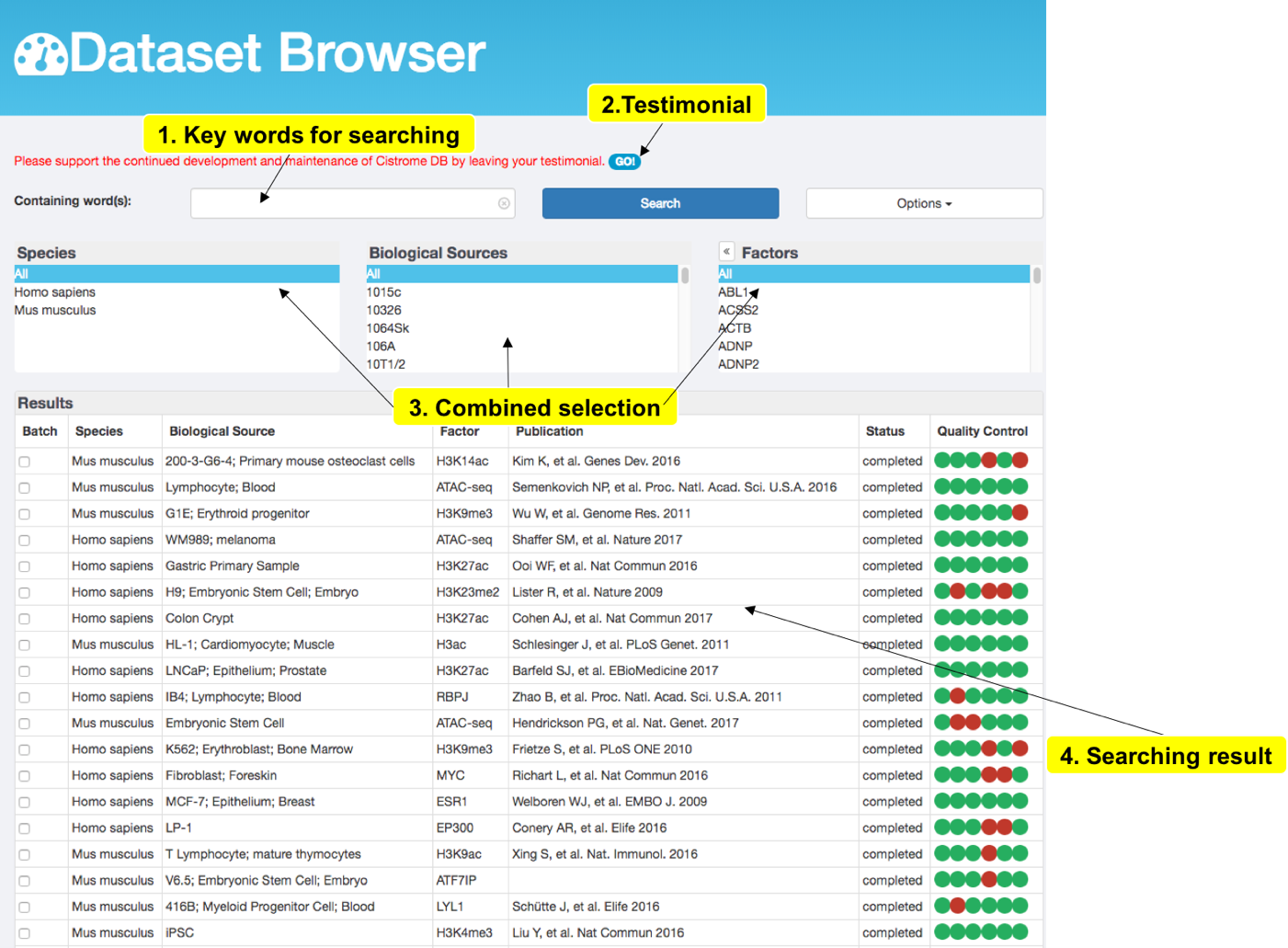
查看某个数据集
点击某个数据集可查看其实验信息, 质量信息,在基因组浏览器查看峰图,下载peak和峰图文件,下载靶基因。
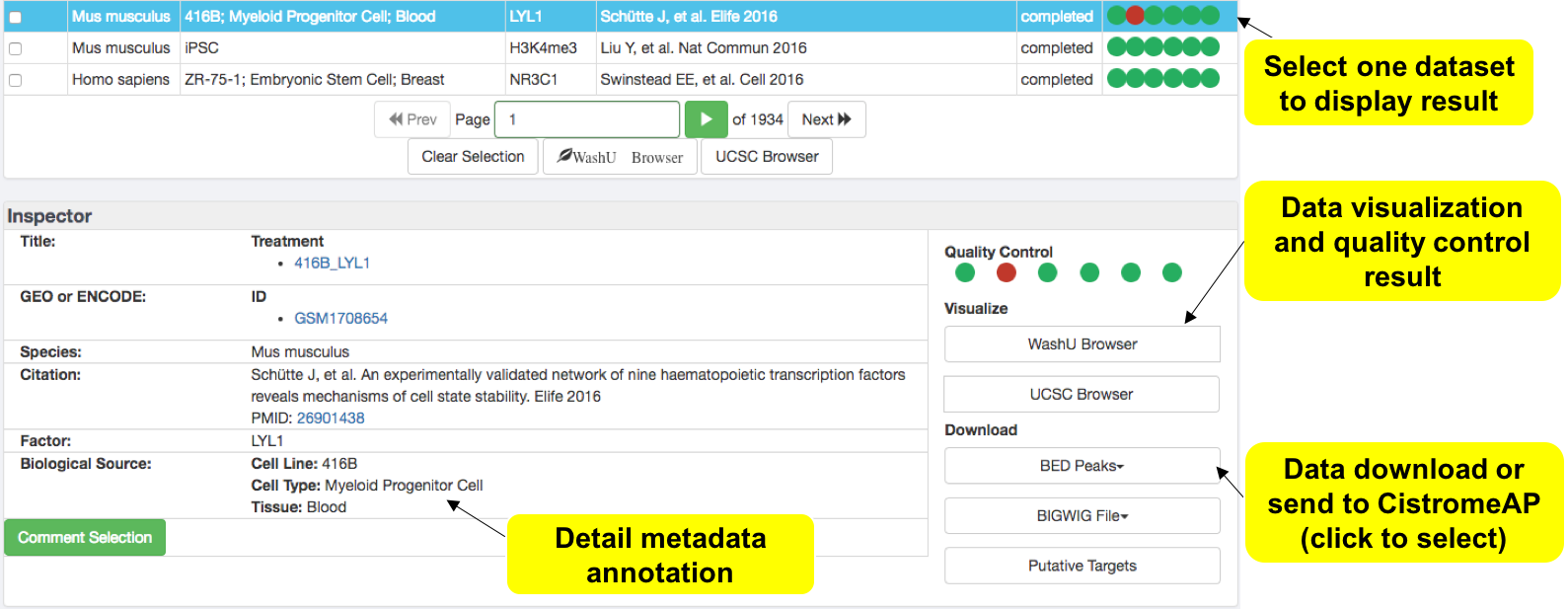
多个样品的靶基因列表下载下来,可以用高颜值在线绘图绘制Venn图,查看交并集情况。
自己处理peak文件,不要忘了可以使用bedtools (简单好上手)。
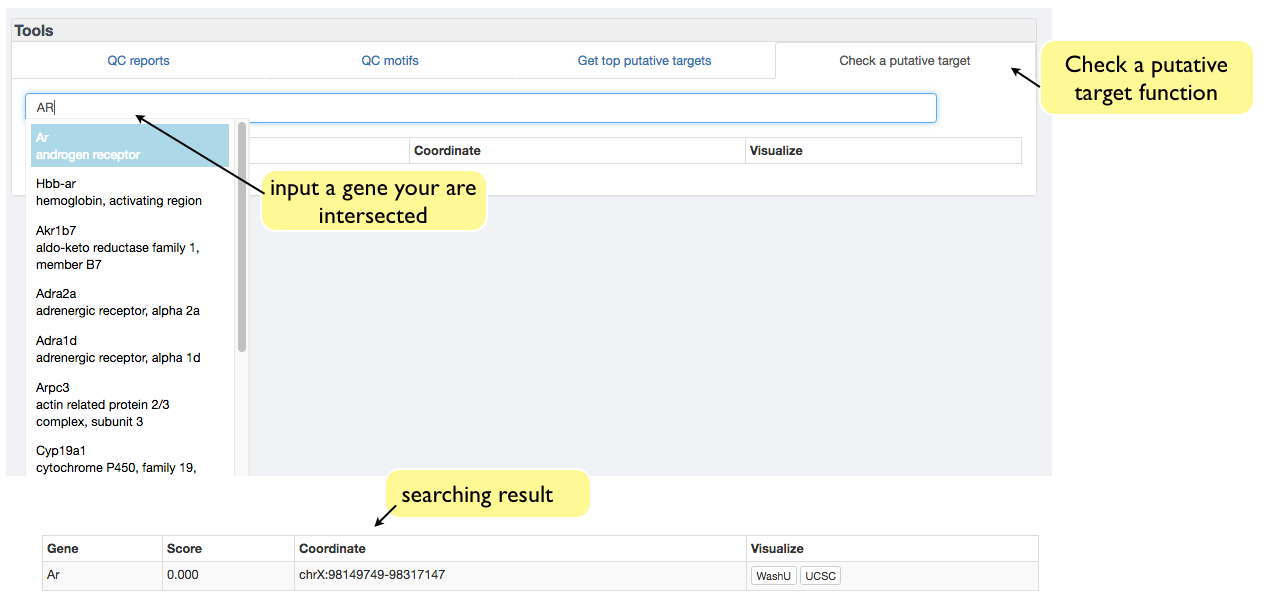
查找目标基因是否受调控
在Check a putative target处输入目的基因,查看其受调控情况,还可以跳转到基因组浏览器,查看更多信息。
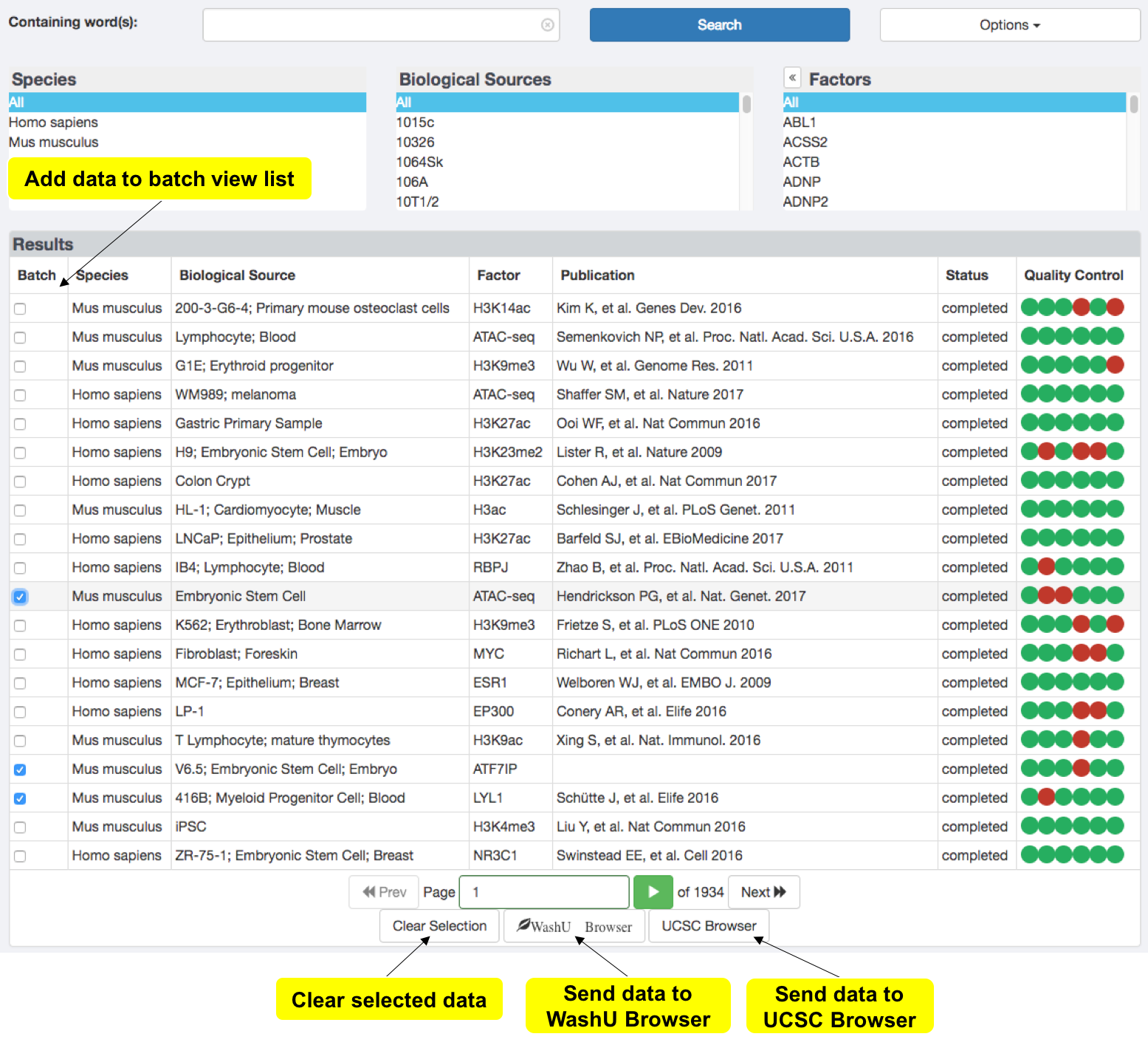
多个样品比较查看
如果关注不同分化水平或不同组织之间转录一直结合、组蛋白修饰水平的差别和靶基因的差别,可同时选中目标样品,一键导入UCSC基因组浏览器或WashU Browser基因组浏览器查看, 生成ChIP类文章不可缺少的reads分布峰图。

数据批量下载
也可以填写申请,批量下载关注的数据, 利用我们提供的分析流程进行进一步的分析。
想不想赶一下大牛的分析
在广大粉丝的期待下,《生信宝典》联合《宏基因组》于2018年4月14在北京鼓楼推出《ChIP-seq分析专题培训》,为大家提供一条走进生信大门的捷径、为同行提供一个ChIP-seq实战分析学习和交流的机会、助力学员真正理解分析原理和完成实战分析, 独创线下集中授课2天+自行练习5天+再集中讲解答疑2天+后期学习群的四段式教学,并提供学习视频,教、学、练、答结合,真正实现独立分析大数据。
关于学习生物信息学分析的重要性,请阅读《生物信息9天速成班—成为团队中不可或缺的人》。
ChIP-seq基本分析流程见流程。课程将在这个基础上,提供更深入地分析指导。
- 座位按报名并成功缴费顺序从前到后龙摆尾式排序。
- 赠送价值188元线上生信基础课程一门,目前的《应用Python处理生物信息数据和作图》、《生物信息作图系列R、Cytoscape及图形排版》和《生物信息中的Linux应用》任选其一。
- 获赠32G品牌定制U盘 (内含数据资料)。
- 多人(N,10>N>1)组团报名并同时缴费,每人还可获得价值N百元的礼品(京东购物卡)。
